2022年10月16日,国际知名学术期刊《核酸研究》(Nucleic Acids Research)在线发表了复旦大学生命科学学院徐书华教授团队、中国科学院上海营养与健康研究所张国庆研究员、复旦大学生命科学学院樊少华研究员合作开发的人类基因组结构变异数据库PGG.SV(https://www.biosino.org/pggsv/),文章题为“PGG.SV: a whole-genome-sequencing-based structural variant resource and data analysis platform”。该数据库通过收集全球人群的全基因组测序数据,专注于基因组结构变异数据的挖掘和整合,为人类基因组结构变异的研究提供了一个数据获取、信息查询和在线分析的综合平台。
基因组结构变异(SVs)主要包括基因组上大片段的DNA缺失、插入、片段重复等变异类型,大量研究表明SV与癌症、自闭症、神经发育障碍等多种复杂遗传病有关,近年来在医学和遗传学领域中持续受到关注。随着基因组测序技术的进步和普及,大量的结构变异被不断发现和研究,一些具有强致病性的结构变异也逐渐得到验证。研究团队旨在通过构建一个具有代表性、多样性的健康人群基因组结构变异数据集,一方面为遗传病患者的结构变异研究提供可靠的对照样本,另一方面对变异功能的注释和预测,也将有效缩小致病性突变的筛选范围,为相关领域研究者提供有效的指导和帮助。
由于结构变异在不同地区和民族之间存在显著差异和多样性,而现有的数据库和公共数据集各自采用不同分析流程,因此一直缺乏一个具有人群样本和新一代测序数据代表性的结构变异资源和分析平台,尤其对东亚人群样本的覆盖度严重不足。研究团队利用对大规模的测序数据的整合,共覆盖了全球177个代表性地区和族群的6,048个全基因组测序样本,特别是对我国丰富的民族多样性特征进行了深度分析,首次覆盖了我国50个少数民族。截至论文发表,数据库共收录了584,277个结构变异,并将在未来持续增加。此外,PGG.SV首次纳入了三代长读长(long-reads)测序数据,其在结构变异的检测中具有更大优势,特别是在插入序列的检测和判定方面,其效果显著优于二代测序技术。先前的大规模结构变异数据库均基于二代测序或基因芯片数据构建。研究团队产生和收集了1,030个三代测序基因组,并首次采用三代测序与二代测序结合的方式构建结构变异数据库,从而大幅提升了结构变异检测结果的数量和质量。
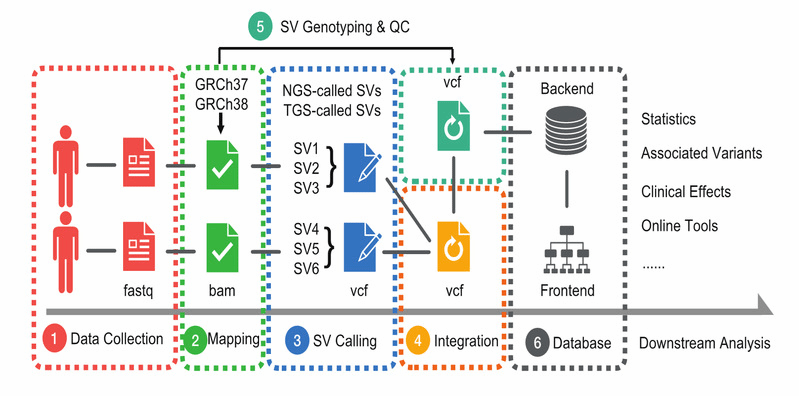
1 PGG.SV数据处理流程示意图
在数据库功能上,PGG.SV提供了简洁友好的查询功能,提供不同族群结构变异在基因组位置上的精确展示,以及全球各个族群之间的频率差异等统计信息。利用课题组先前积累的优势,PGG.SV与徐书华教授团队此前开发的PGG.SNV等数据库进行联动,借助连锁不平衡和基因组空间位置信息,将单核苷酸变异(SNV)的详细结果与结构变异相结合,以增强数据多样性的解析功能。此外,PGG.SV提供了丰富的临床效应分析和预测分析功能,根据与结构变异存在关联的基因和调控元件,提供对其潜在表型、功能的预测和富集分析,以及由特定疾病和表型检索相关结构变异的工具,以便有临床研究等需求的用户使用。
最后,PGG.SV还支持丰富的在线分析和可视化功能。一方面,研究团队提供对用户提交的结构变异结果的比较和注释,以便使用者了解自己的目标样本与数据库提供的对照样本之间的差异;另一方面,研究团队还提供结构变异可视化功能,能够在人类基因组上检索用户提交的DNA序列、展示相关变异的基因组位置,以及提供对变异空间结构变化的精细可视化。
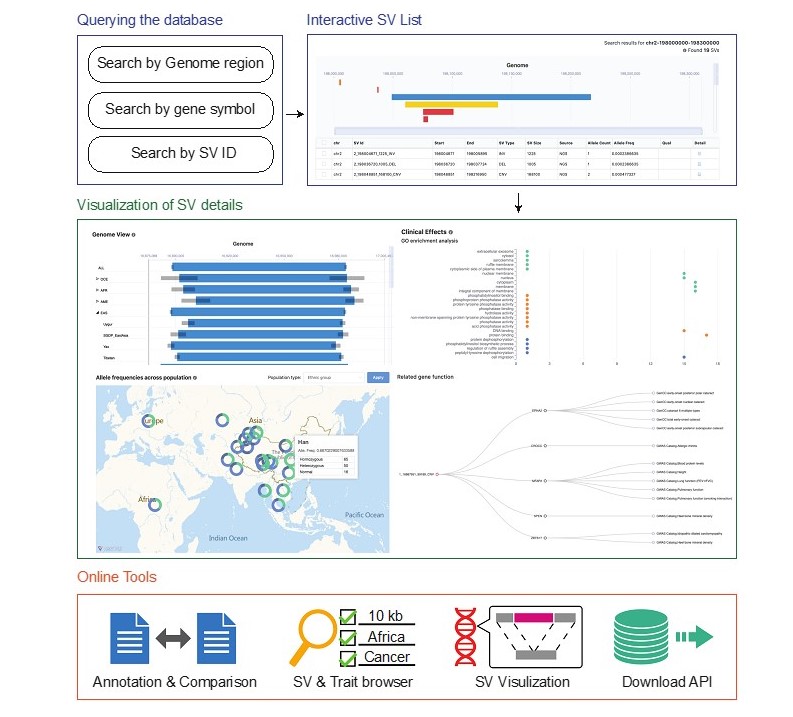
2 PGG.SV界面示意图
总体而言,PGG.SV提供了一个高质量的人群基因组结构变异数据资源,基于新一代测序数据对人类基因组结构变异信息检测和展示进行了大幅度提升,尤其是首次较为全面地覆盖了东亚人群和中国人群的结构变异多样性,并提供相关基因和潜在临床效应的注释。此外,该平台也提供了包括病例对照研究在内的多种在线分析功能,以及人类基因组结构变异的可视化工具。
中国科学院上海营养与健康研究所王亦民、凌鋆超和复旦大学人类表型组研究院龚娇为本文共同第一作者。徐书华教授、张国庆研究员和樊少华研究员为本文共同通讯作者。该研究工作得到了国家基金委基础科学中心、国家自然科学基金、中国科学院先导专项、英国皇家学会牛顿基金、人类表型组上海市市级重大专项等基金的资助。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkac905/6761741?login=false
