2024年8月15日,国际知名学术期刊International Journal of Biological Macromolecule(IJBM)发表了复旦大学徐书华教授和内蒙古大学左永春教授团队题为"Review and new insights into the catalytic structural domains of the Fe(ll) and 2-Oxoglutarate families"的综述性论文。该论文通过序列比较和结构拟合,综述了内大联合团队关于二价铁与2-氧代戊二酸家族KDM、ALKBH和TET蛋白具有相似的催化结构域却具有不同的底物结合偏好性的原因。结果表明KDM、ALKBH和TET蛋白中都存在10个相对保守的β折叠和3个具有明显结构差异的关键区域Region1/2/3,关键区域的差异是其具有不同底物选择性的原因,这促进了人们对去甲基化蛋白家族底物选择性的理解,并为生物技术的进一步研究提供了见解。
组蛋白赖氨酸去甲基化酶(KDM)、AlkB 同源物(ALKBH)和 Ten-Eleven 易位(TET)蛋白隶属于Fe(II)和α-酮戊二酸(2OG)依赖的双加氧酶家族。它们都含有一个以双链 β-螺旋为中心的催化结构域,其拓扑结构限制了直接参与底物结合的区域,致使其具有不同的催化功能。然而,深层结构的生物学原因尚不清楚,这限制了人们对底物选择性抑制开发和生物工程的研究。
人、鼠、牛、羊四个物种在KDM(2-8),ALKBH与TET共计三个蛋白质家族,33亚家族成员的催化结构域特征被归纳。相比于KDM,ALKBH与TET的催化结构域具有更紧密的系统发育关系和更相似的底物类型。KDM,ALKBH与TET蛋白都具有相对保守的10个β折叠和3个差异较大的区域:Region1/2/3。Fe(II) 和 2OG 结合位点夹在两个 β 折叠片之间。相对保守的 HXD/E…H 基序,可与 Fe(II) 结合。各家族成员内的α-KG 结合基序相对保守(图1)。
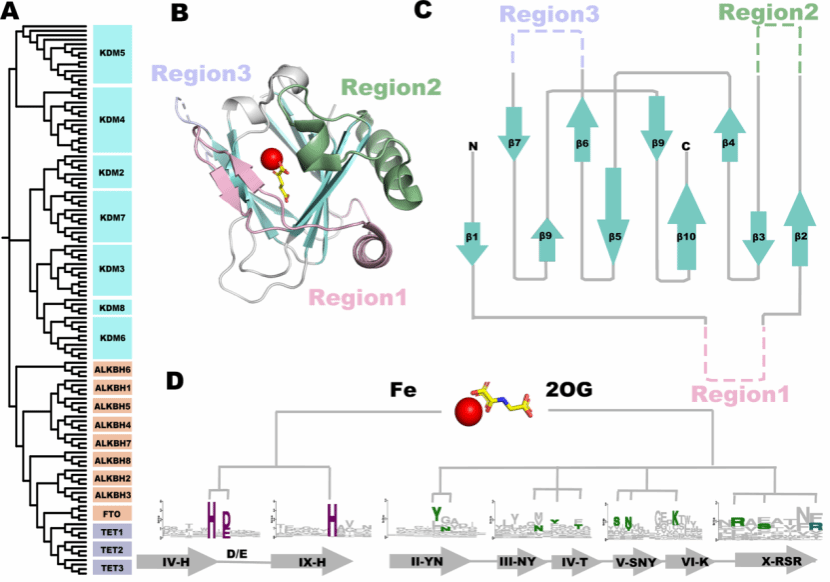
图1.三个蛋白质家族的催化结构域的进化关系和结构特征
三个关键Region在KDM、ALKBH与TET蛋白结合底物的过程中发挥着重要作用。由Flip1和Flip2组成的核酸识别盖(NRL)和NRL外的Flip3是AlkB家族独有的特征,其作为ALKBH成员之间的结构变异而存在。实验研究证明其在底物相互作用中起关键作用,但其并未被系统的总结和归纳。值得注意的是,Flip1/2/3位于该文所确定的Region1/2/3中(图2)。
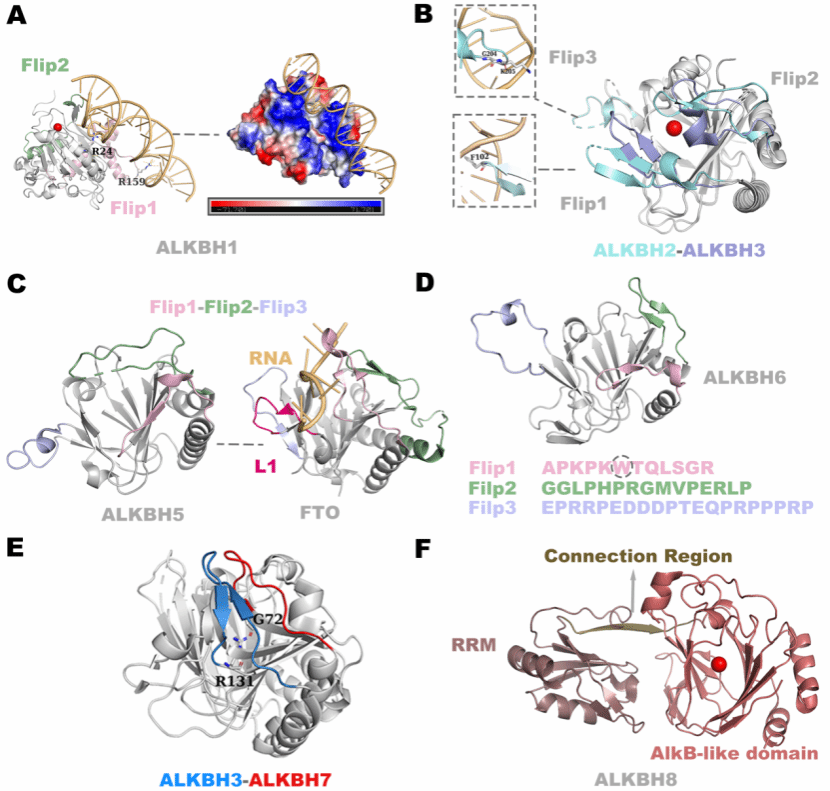
图2.ALKBH蛋白关键区域在底物结合中的作用
此外,Region1/2/3在KDM蛋白和TET蛋白的底物结合作用也被系统的总结。该文为实验学科的研究提供了宝贵的原始数据收集,为KDM2B、ALKBH4、TET1/3等催化结构域结构尚未解析的成员提供了新的研究视角。
该文第一作者杨斯奇是复旦大学生命科学学院徐书华教授和内蒙古大学生命科学学院左永春教授共同指导的博士生。中国科学院大学博士生刘东阳,内蒙古大学研究生邢吉祥、宋彦成,本科生于皓宇对该工作的开展和完成做出了贡献。该项目获得了国家自然科学基金、内蒙古自治区自然科学基金等多项基金的资助。
论文链接:https://www.sciencedirect.com/science/article/abs/pii/S0141813024056034
